Forschungsschwerpunkte
Antimikrobielle Resistenz (AMR) und neue antimikrobielle Strategien
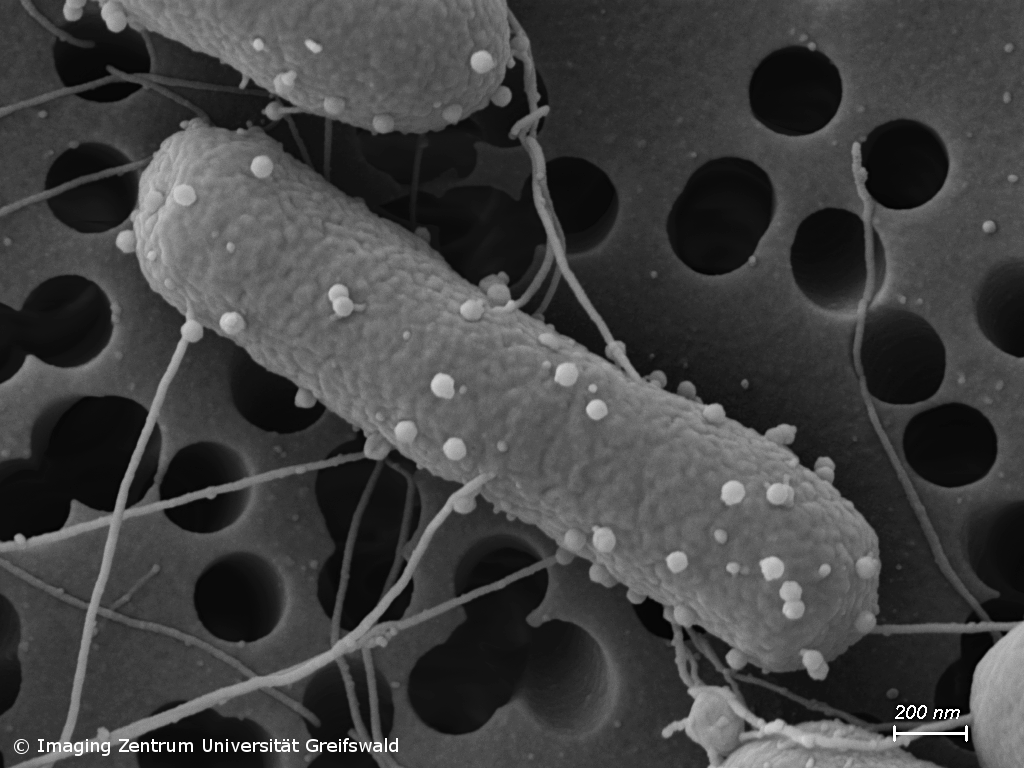
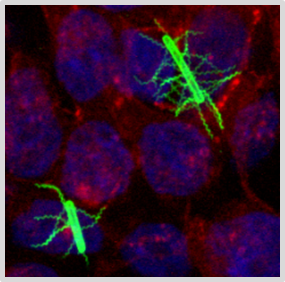
Infektionen mit antimikrobiell resistenten Erregern stellen eine der größten Bedrohungen für die Menschheit dar. Kliniker haben bereits heute mit Bakterien zu kämpfen, die Resistenzen gegen alle derzeit verfügbaren Antibiotika aufweisen. Unsere Gruppe untersucht das Vorkommen und die Übertragung von Erregern mit AMR-Genen, beispielsweise in Abwässern und Böden, und forscht darüber hinaus an der Entwicklung alternativer antimikrobieller Therapien. So analysieren wir beispielsweise die Aktivität und Wirkungsweise neuartiger synthetischer und natürlicher Substanzen gegen problematische bakterielle Erreger und suchen zudem nach Bakteriophagen, die das Potenzial haben, für einen zukünftigen Einsatz in der Phagentherapie weiterentwickelt zu werden.
Pathophysiologie von Clostridioides difficile (CDinfect)
Das streng anaerobe Bakterium C. difficile ist einer der problematischsten Erreger nosokomialer und rezidivierender Infektionen. Der Darmpathogen kann im Darm überleben und Infektionen auslösen, da er hochresistente Endosporen bildet und den rauen Bedingungen des menschlichen Darmtrakts standhält. Trotz der Bedeutung von C. difficile-Infektionen ist die Physiologie des Erregers noch immer unzureichend verstanden. Unser Ziel ist es, die wichtigsten Stoffwechselwege und physiologischen Mechanismen zu identifizieren, die es C. difficile ermöglichen, im Darmtrakt zu gedeihen und schwere Infektionen beim Menschen zu verursachen.
Oxidativer Stress und antioxidative Strategien (BiOx GRK1947, FlavOx DFG)
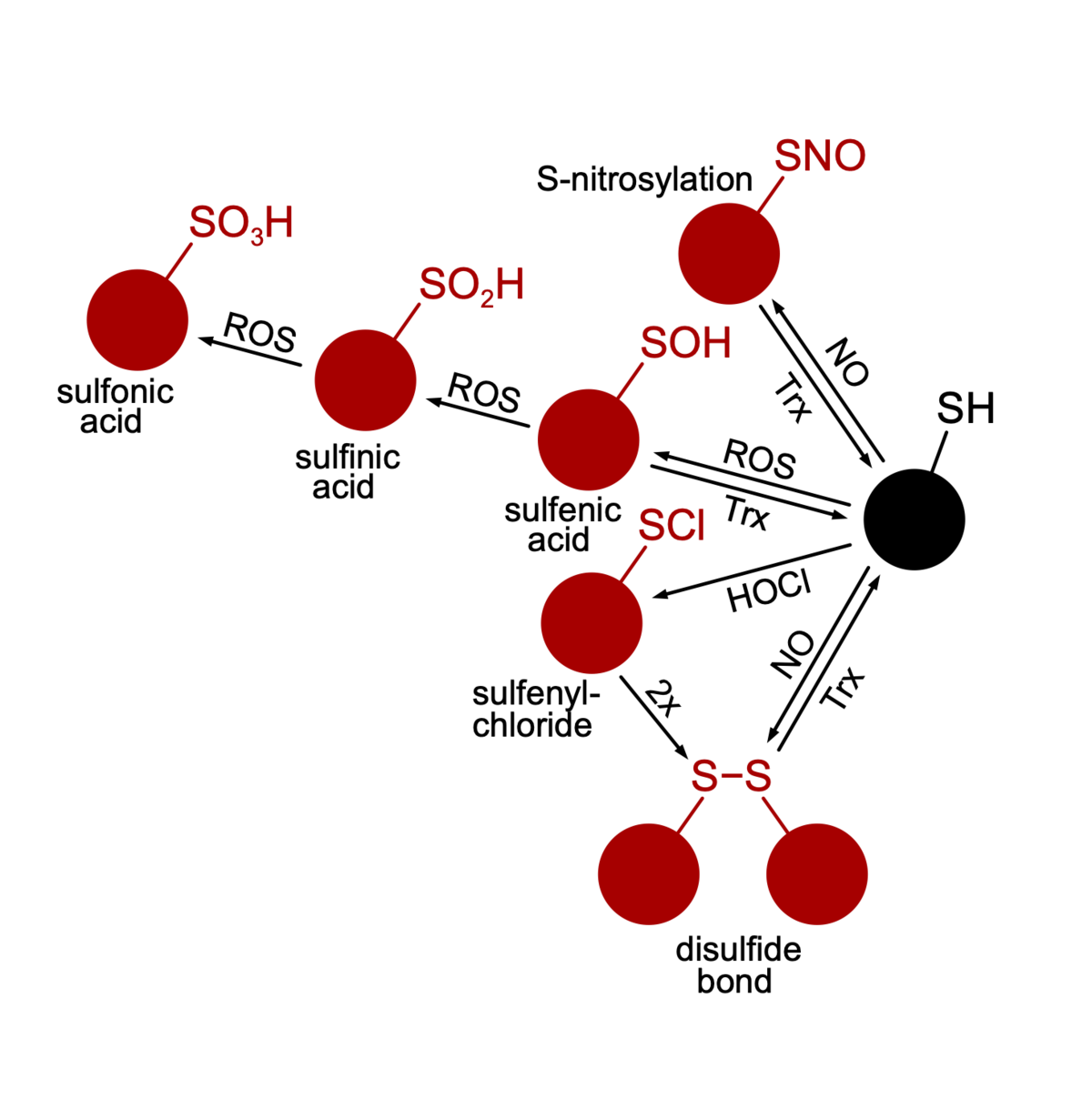
Das Leben basiert auf Redoxreaktionen, d. h. die Zelle ist voll von Molekülen, die sich gegenseitig reduzieren oder oxidieren. Einige Moleküle haben ein größeres oxidatives Potenzial als andere. Vor allem in Gegenwart von Sauerstoff entstehen reaktive Sauerstoffspezies (ROS), die das Potenzial haben, zelluläre Strukturen zu oxidieren und dadurch Signalübertragungswege zu initiieren oder Makromoleküle zu schädigen. Mikroorganismen, die in ihrem Stoffwechsel auf Sauerstoff angewiesen sind, verfügen über ausgeklügelte Stressreaktions-, Reparatur- und ROS-Entgiftungsmechanismen. Man könnte annehmen, dass streng anaerobe Organismen über solche Mechanismen nicht verfügen, da sie in sauerstofffreien Lebensräumen wachsen. Viele streng anaerobe Bakterien sind jedoch zu einer oxidativen Stressreaktion fähig und können daher kurze Zeiträume mit Sauerstoff und ROS überleben. Bei anaeroben Krankheitserregern kann die Fähigkeit, Perioden von oxidativem Stress zu überleben, darüber entscheiden, ob sich der Erreger im Wirt durchsetzen kann oder nicht. Wir untersuchen die oxidative Stressantwort in C. difficile und klären dabei ROS-Sensoren, Signaltransduktionswege, Schutzmoleküle und Reparaturmechanismen auf. Ein besonderer Schwerpunkt liegt dabei auf oxidierten Proteinen. Mit Hilfe von Massenspektrometrie-basierter Proteomik und Redox-Proteomik wollen wir Redox-Schalter und besonders anfällige Strukturen in der C. difficile-Zelle aufspüren.
Membranstruktur und membranaktive Verbindungen
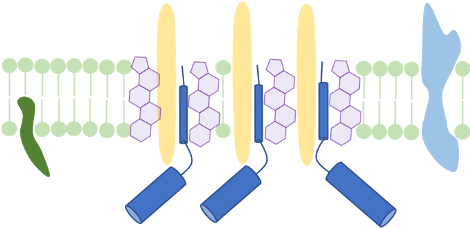
Die Zellmembran trennt den intrazellulären Raum vom Außenraum. Sie gewährleistet nicht nur die Zellintegrität, sondern ermöglicht auch Transport-, Signalempfangs- und -transduktionsprozesse. Wir nutzen Membranproteomik und mikroskopische Techniken, um den Einfluss antimikrobieller Substanzen auf Membranstruktur und -funktion zu untersuchen. Aktuell analysieren wir Veränderungen funktioneller Membranmikrodomänen in C. difficile in Gegenwart infektionsrelevanter Gallensäuren. Wir gehen davon aus, dass membranaktive Substanzen aus dem Wirt oder Mikrobiom eine kombinatorische Wirkung mit antimikrobiellen Substanzen haben könnten, die therapeutisch bei Infektionen eingesetzt werden.
Bodenmikrobiom und seine Auswirkungen auf landwirtschaftliche Nutzpflanzen (AgriBiom T!Raum One Health BMBF)

Es gibt praktisch keinen Ort auf der Erde, der frei von Mikroben ist. Spezifische Nischen verfügen über eine eigene mikrobielle Gemeinschaft – ein nischenspezifisches Mikrobiom, dessen Rolle für die Stabilität und das Gleichgewicht dieser Nische oft noch unbekannt ist. Unsere Expertise in massenspektrometriebasierter Proteomik und Metaproteomik ermöglicht uns detaillierte Analysen des Bodenmikrobioms landwirtschaftlicher Flächen. Unser Ziel ist es einerseits, die Mikroben und ihre Stoffwechselfähigkeiten zu entschlüsseln, die für Wachstum und Gesundheit landwirtschaftlicher Nutzpflanzen förderlich sind, und andererseits mikrobielle Parameter zu identifizieren, die auf schädliche Auswirkungen des Bodenmikrobioms hinweisen.
Aktuelle Projekte
- T!Raum One Health
AgriBiom "Etablierung einer Standard-Diagnostik des Mikrobioms agrikultureller Böden"
Mai 2026 - Erforschung der Ökologie und Evolution von Phagen, um ihren potenziellen Nutzen für den Menschen aufzudecken (UtiliPhage)
März 2027
Abgeschlossene Projekte
- BIOx (FKZ GRK 1947): Biochemische und biophysikalische Effekte reaktiver Sauerstoff- und Stickstoffspezies auf biologische Membranen,
TP C4: “The membrane of the intestinal pathogen C. difficile as the first line of defense during infection-related oxygen and bile acids challenge” - FlavOx (FKZ SI 1678/3-1): Aufklärung der Bedeutung von Flavodoxinen im intestinalen Pathogenen Clostridioides difficile
- Molekulare Grundlagen der Wirtsadaption und Pathogenität zoonotischer Hantaviren (HANTadapt)
Arbeitsgruppe "Datenanalyse und Datenintegration" (Dr. Jörg Bernhardt)
Sehr große Datensätze werden vor allem durch "Omics" Technologien und Meta-Studien generiert. Wir arbeiten an globalen Analyse-Strategien und wie man Daten aus verschiedenen "Omics" Technologien wie Transcriptomics, Proteomics und Metabolomics integriert. Dadurch können neue Einsichten in die adaptive Physiologie und Pathogenität von Bakterien (z. B. Staphylococcus aureus) gegeben werden. Darüber hinaus interessieren wir uns für komplexe mikrobielle Gemeinschaften (Mikrobiome) und arbeiten an geeigneten Metaanalyse-Arbeitsabläufen. Prophane (http://www.prophane.de) und Aureolib (http://aureolib.de) sind Beispiele für erfolgreiche Datenanalyse bzw. Integrationsquellen, die von unserer Gruppe initiiert wurden.
Arbeitsgruppe „Stress Physiology" (Dr. Ulf Gerth)
- Die σB-abhängige generelle Stressantwort von Bacillus subtilis:
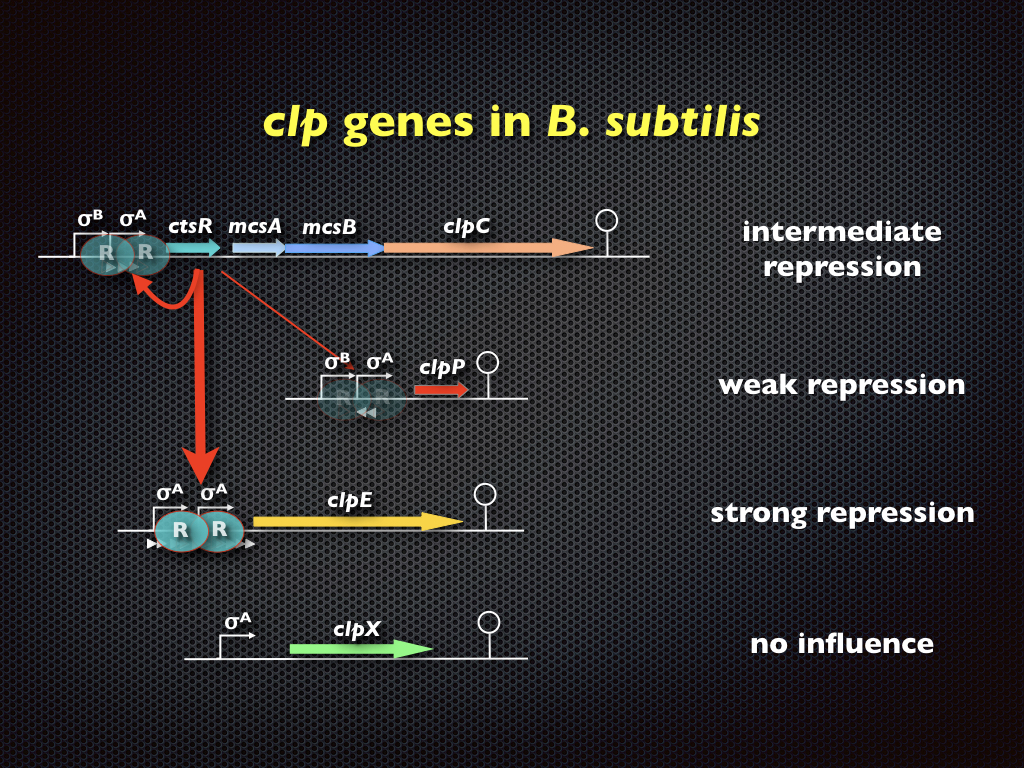
Mehr als 200 Gene gehören zum σB-abhängigen generellen Stressregulon. Diese Proteine statten nicht wachsende Zellen mit einer multiplen, unspezifischen und präventiven Stressresistenz in Erwartung von "zukünftigem Stress" aus. Die "Feinabstimmung" der Genregulation und die Funktion einzelner Stressproteine bei der Etablierung einer globalen Antwort auf Hitze, Ethanol, oxidativen und osmotischen Stress werden derzeit analysiert. Darüber hinaus wird die Integration der generellen Stressantwort in ein hochentwickeltes Adaptionsnetzwerk untersucht. - Die Bacillus subtilis "Clp Maschinerie”:
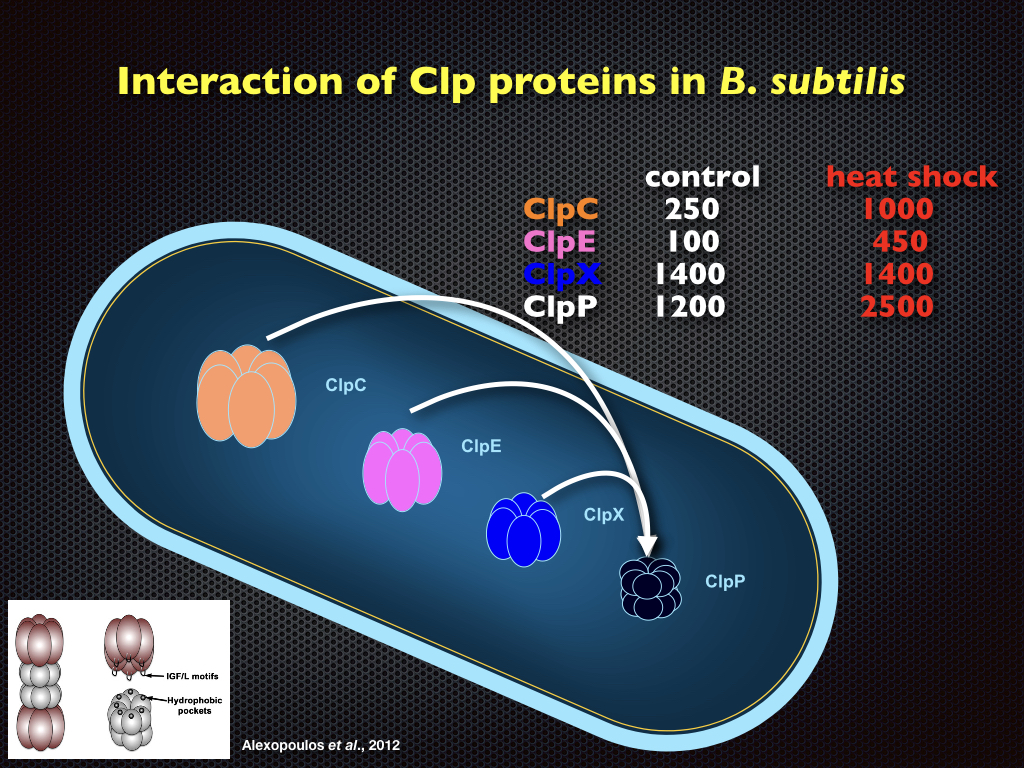
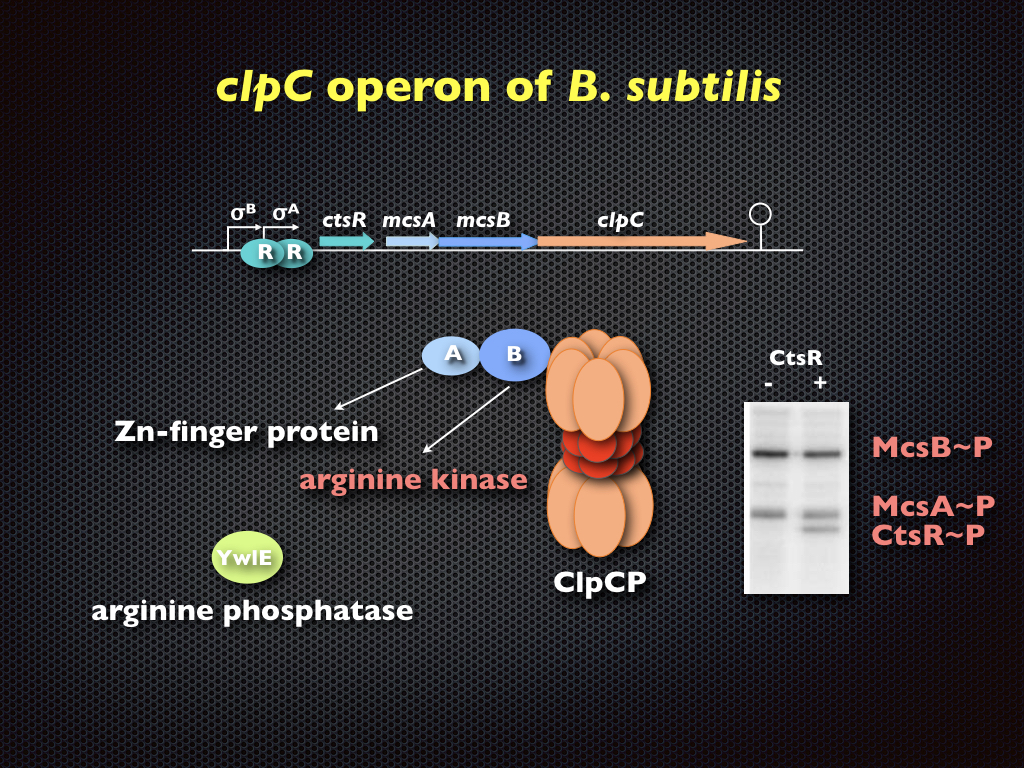
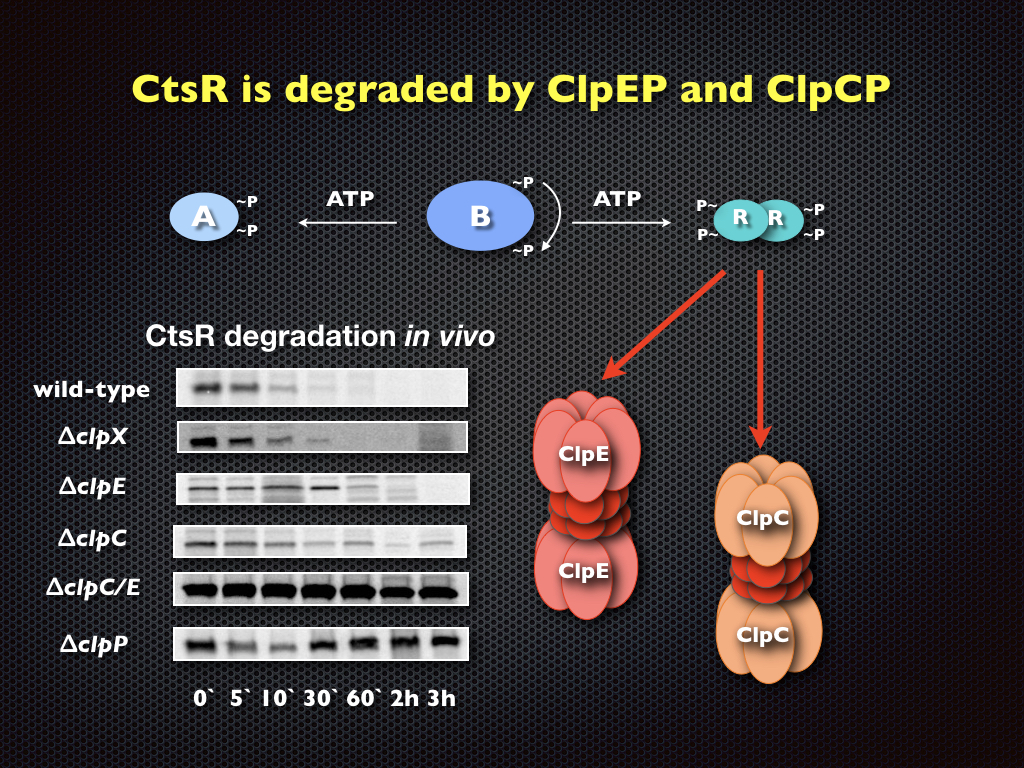
Die Expression der stress-induzierbaren clp Gene hängt vorwiegend vom CtsR Transkriptions-Repressor ab. CtsR wird durch die ClpEP- und ClpCP-Proteasen nach Hitzeschock abgebaut. Darüber hinaus kann die ATP-abhängige Proteolyse, die durch Clp-Proteasen vermittelt wird, auch bei allgemeinen Phänomenen der stationären Phase beobachtet werden, wie Glukose-Hunger, Kompetenzentwicklung und Sporulation. Mechanismen, die schließlich zur Inaktivierung und Abbau von Transkriptionsregulatoren und Proteinen führen, die ihre "Pflicht" getan haben, werden derzeit untersucht. Darüber hinaus steht die Rolle der McsB-Argininkinase, ein Adaptorprotein der ClpCP-Protease, im Mittelpunkt unserer Forschung.
.
Weitere Schlüsselpublikationen:
- Reder et al. Cross-talk between the general stress response and sporulation initiation in Bacillus subtilis – the σB promoter of spo0E represents an AND-gate. Environ Microbiol (2012) 14(10), 2741–2756. PMID: 22524514.
- Reder et al. The modulator of the general stress response, MgsR, of Bacillus subtilis is subject to multiple and complex control mechanisms. Environ Microbiol (2012) 14(10), 2838–2850.PMID: 22812682.
- Elsholz et al. CtsR, the Gram-positive master regulator of protein quality control, feels the heat. EMBO J. 2010 Nov 3;29(21):3621-9.
- Elsholz et al. Global impact of protein arginine phosphorylation on the physiology of Bacillus subtilis. PNAS 2012 May 8;109(19):7451-6 PMID: 22517742.